藜麦(Chenopodium quinoa)被印加帝国称为“粮食之母”。大约7,000年前,藜麦被前哥伦布人驯化。藜麦种子含有极高的营养价值,因为它不含麸质,含糖量低,各营养物质,如氨基酸、纤维、碳水化合物、矿物质、维生素等达到完美的平衡。同时,藜麦的抗逆性也很强,能适应多种恶劣的生长环境。藜麦基因组较复杂,为异源四倍体(2n=4x=36),估计的基因组大小为~1.5Gb。
2016年日本科学家在DNA research (IF: 5.267)发布了藜麦的一张基因组图谱。采用的是高覆盖度的二代(Illumina Hiseq 2500; ~200 x)+低覆盖度的三代(PacBio RS II; ~30 x)的测序策略仅得到藜麦基因组的草图,组装得到大约25,000个scaffold。
本文作为封面文章,于今年发表于Nature(IF: 28.138)杂志。本文是来自沙特阿卜杜拉国王科技大学的研究人员与多国研究人员合作,采用纯三代数据结合Bionano光学图谱、Dovetail Hi-C染色质互做数据和遗传图谱的组装测序,得到了染色体级别的藜麦基因组精细图。
研究内容与结果
1. 测序策略和组装结果
利用纯三代测序,藜麦的contig N50达到了1.6 Mb。结合Bionano光学图谱和Dovetail Hi-C的数据,藜麦基因组的scaffold N50达到了~3.8Mb,scaffold的数目仅为~3,400个。
藜麦是由祖源的两个二倍体品种(亚基因组A和B)杂交而来。之前的单基因测序研究发现北美和欧亚的两个二倍体分别为A亚基因组和B亚基因组的候选来源。二者可能在北美某处发生了杂交。为了进一步了解藜麦的进化历史,作者对二倍体亚基因组A(C. pallidicaule)和二倍体亚基因组B(C. suecicum)也进行了测序和组装。
2. 藜麦的进化历史
藜麦中有很大比例的同源基因具有相似的同义替代率,表明藜麦基因组中可能存在全基因组的复制事件。而这种现象在亚基因组A和B中是不存在的。根据同义同义替代率和拟南芥以及真核生物的突变率,作者估计亚基因组A和B杂交形成藜麦的时间约在3.3-6.3百万年前。

在亚基因组A和B杂交后,其实形成了多个可相互杂交的四倍体物种,包括C. berlandieri和C. hircinum。为了进一步确定藜麦与二倍体的亚基因组A和B以及这些四倍体近缘物种之间的进化关系,作者挑选了15个藜麦品种进行重测序。这些品种代表了藜麦的两种主要生态型:高原和海滨品种。对这些品种进行进化树分析表明北美C. berlandieri可能是这些种的基本成员。以前的研究认为藜麦是由C. hircinum经单次驯化而来。而本文的进化树分析则表明藜麦可能是在高原和海滨环境下分别独立驯化而来。

3. 亚基因组特点分析
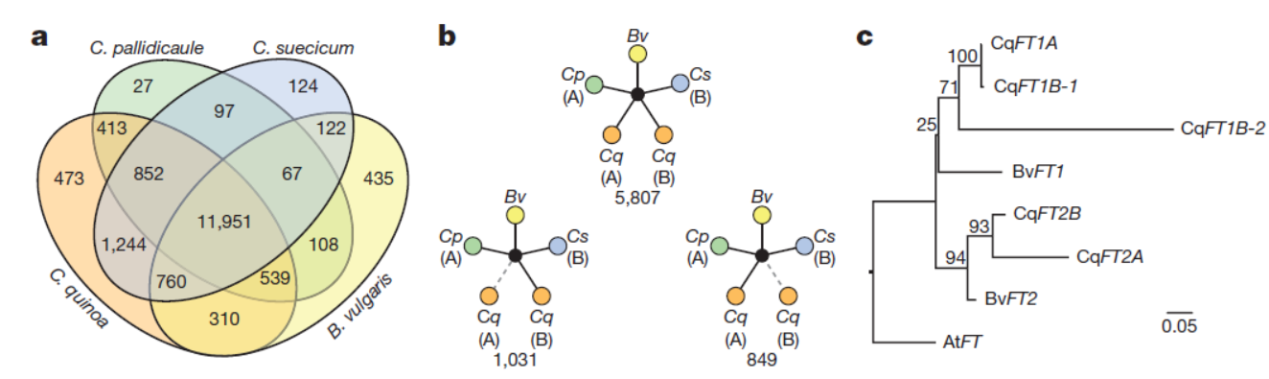
利用BLASTN将亚基因组A (C. pallidicaule)和B(C. suecicum)的序列与藜麦的scaffold比对,发现分别有156个和410个藜麦scaffold比对到A基因组和B基因组上(总共202.6Mb和646.3Mb)。根据亚基因组A和B中5,807个同源基因对的序列比对信息,发现二者高度同源。仔细研究也发现了两个亚基因组中存在重组、染色体重排和染色体融合的现象。

利用OrtholMCL对藜麦和两个亚基因组进行基因家族分析。有5,807个单拷贝基因仍保留在亚基因组杂交后,而分别有1,031和849个单拷贝在两个亚基因组A和B中消失。作者也对多拷贝基因进行了分析。比如,在B. vulgaris中有两个拷贝的FT基因。而在藜麦中发现有6个拷贝的FT基因。通过对基因序列的比较,发现在藜麦中该基因可能存在局部tandem duplication的现象。
4. 皂角苷形成的分子机制
皂角苷是藜麦种子中苦味的来源,同时也可能导致人类食用时的溶血。因此,藜麦育种倾向于培育无皂角苷的品种——甜味藜麦。为了弄清楚皂角苷形成的分子机制,作者构建了甜味藜麦和苦味藜麦杂交的分离群体,对后代做了BSA(Bulked Segregant Analysis)性状定位和连锁定位。CqB16的scaffold 3489被鉴定为候选区域。通过对定位候选区域内基因功能和结构的分析,TSARL1被认为是控制皂角苷形成的候选基因。
参考文献:
Yasui Y, Hirakawa H, Oikawa T, et al. Draft genome sequence of an inbred line of Chenopodium quinoa, an allotetraploid crop with great environmental adaptability and outstanding nutritional properties[J]. DNA Research, 2016, 23(6):535-546.
Jarvis DE,Ho YS, Lightfoot DJ,et al. The genome of Chenopodium quinoa. Nature, 2017, 16;542(7641):307-312.